Oferecido por
A explosão dos modelos de Inteligência Artificial (IA) de conversão de texto em imagem, (programas treinados para gerar imagens de quase tudo o que você pedir) como o DALL-E 2 da OpenAI, repercutiu em diversos segmentos das indústrias criativas, da moda ao cinema, pela sua capacidade de fornecer imagens estranhas e maravilhosas sob demanda.
A mesma tecnologia por trás desses programas também está fazendo sucesso nos laboratórios de biotecnologia, que começaram a usar esse tipo de IA generativa, conhecida como modelo de difusão (diffusion models), para criar moldes para novos tipos de proteína nunca vistos antes na natureza.
Em dezembro, dois laboratórios anunciaram separadamente programas que usam modelos de difusão para gerar moldes para novas proteínas com mais precisão do que nunca. A Generate Biomedicines, uma startup com sede em Boston (EUA), apresentou um programa chamado Chroma, que a empresa descreve como o “DALL-E 2 da biologia”.\
No mesmo mês, uma equipe da Universidade de Washington (EUA) liderada pelo biólogo David Baker construiu um programa semelhante chamado RoseTTAFold Diffusion. Em um artigo pré-impresso publicado online no dia 1º de dezembro, Baker e seus colegas mostram que seu modelo é capaz de gerar padrões precisos para novas proteínas que podem ser desenvolvidas em laboratório. “Estamos criando proteínas sem nenhuma semelhança com as existentes”, diz Brian Trippe, um dos co-desenvolvedores do RoseTTAFold.
Esses programas podem ser instruídos para produzir padrões de proteínas com propriedades específicas, como forma, tamanho ou função. Com efeito, isso possibilita a criação sob demanda de novas proteínas para realizar trabalhos específicos. Os pesquisadores esperam que isso acabe levando ao desenvolvimento de medicamentos novos e mais eficazes. “Podemos descobrir em minutos o que levou milhões de anos para se desenvolver”, diz Gevorg Grigoryan, CTO da Generate Biomedicines.
“O que é marcante neste trabalho é a geração de proteínas de acordo com as restrições desejadas”, diz Ava Amini, biofísica da Microsoft Research em Cambridge, Massachusetts (EUA).
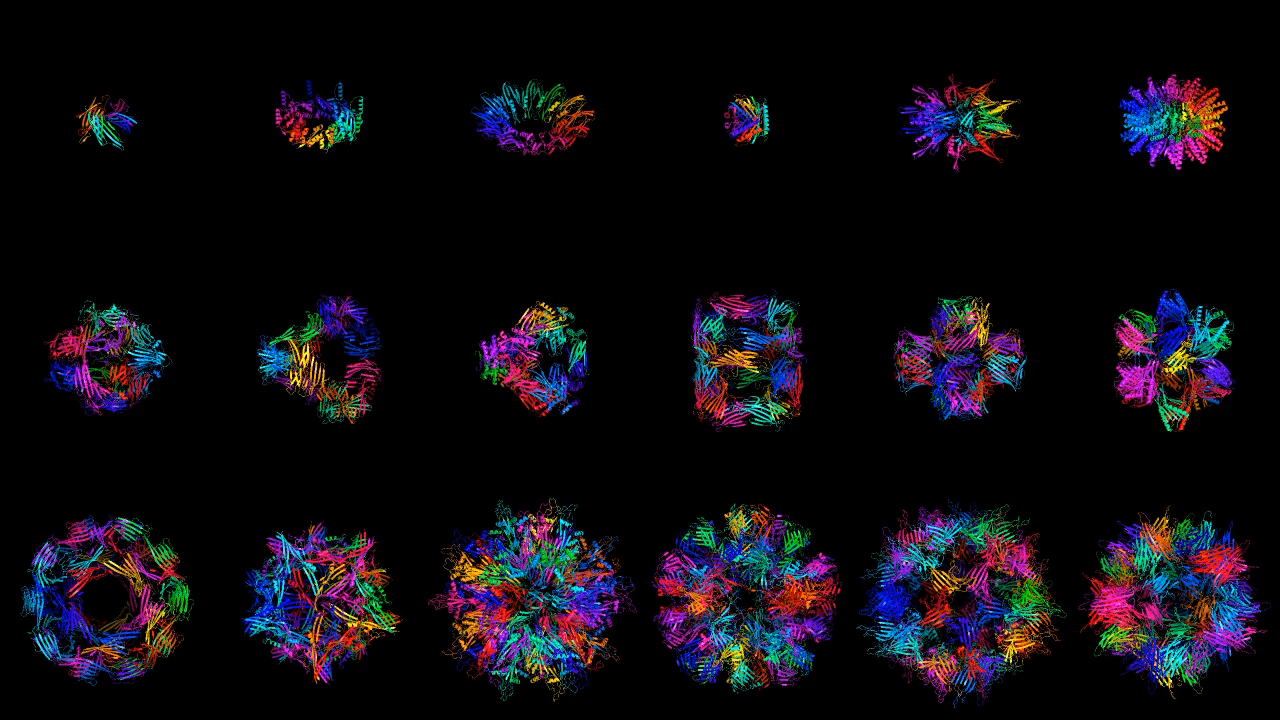
As proteínas são os pilares fundamentais dos sistemas com vida. Nos animais, elas digerem alimentos, contraem músculos, detectam luz, orientam o sistema imunológico e muito mais. Quando as pessoas ficam doentes, as proteínas desempenham um papel ativo em resposta.
Portanto, elas são os principais alvos dos medicamentos. E, atualmente, muitos dos mais novos já são baseados em proteínas. “A natureza usa proteínas para praticamente tudo”, diz Grigoryan. “A esperança que elas oferecem para intervenções terapêuticas é realmente imensa”.
Mas hoje os fabricantes de medicamentos precisam recorrer a uma lista de ingredientes composta de proteínas naturais. O objetivo da geração de proteínas por IA é aumentar essa lista com um conjunto quase infinito de proteínas projetadas por computador.
Técnicas computacionais voltadas para isso não são novas. Mas as abordagens anteriores foram lentas e não muito boas em criar grandes proteínas ou complexos de macromoléculas orgânicas, que atuam como máquinas moleculares compostas de múltiplas proteínas acopladas. E elas são muitas vezes cruciais para o tratamento de doenças.
Os dois programas anunciados no dia 1º de dezembro também não são os primeiros a usar modelos de difusão para geração de proteínas. Um punhado de estudos nos últimos meses de Amini e outros cientistas mostraram que os modelos de difusão são uma técnica promissora, mas eram apenas protótipos de prova de conceito. Chroma e o RoseTTAFold Diffusion se baseiam nesses trabalhos e são os primeiros programas completos que podem produzir designs precisos para uma ampla variedade de proteínas.
Namrata Anand, que co-desenvolveu um dos primeiros modelos de difusão para geração de proteínas em maio de 2022, acredita que o grande significado do Chroma e do RoseTTAFold Diffusion é que eles pegaram a técnica e a superdimensionaram, treinando os programas com mais dados e mais computadores. “Pode ser justo dizer que eles são mais parecidos com o DALL-E por causa de como eles expandiram as coisas”, diz ela.
Os modelos de difusão são redes neurais treinadas para remover “ruído” (perturbações aleatórias adicionadas aos dados) de seu input. A partir de uma confusão aleatória de pixels, um modelo de difusão tentará transformar os dados em uma imagem reconhecível.
No Chroma, o ruído é agregado ao desvendar as cadeias de aminoácidos das quais uma proteína é feita. Com um aglomerado aleatório dessas cadeias, o Chroma tenta juntá-las para formar uma proteína. Em seguida, guiado por restrições específicas sobre como o resultado deve ser, ele pode gerar novas proteínas com propriedades específicas.
A equipe de Baker adota uma abordagem diferente, embora os resultados finais sejam semelhantes. Seu modelo de difusão usa uma estrutura inicial ainda mais embaralhada. Outra diferença fundamental é que o RoseTTAFold Diffusion usa informações sobre como as partes de uma proteína se encaixam por meio de uma rede neural separada treinada para prever a estrutura da proteína (como o AlphaFold da DeepMind faz). Isso serve como orientação para o processo generativo geral.
A Generate Biomedicines e a equipe de Baker mostram uma impressionante variedade de resultados com os programas. Eles são capazes de gerar proteínas com vários graus de simetria, incluindo proteínas circulares, triangulares ou hexagonais. Para ilustrar a versatilidade de seu programa, a Generate Biomedicines criou proteínas com o formato das 26 letras do alfabeto latino e dos numerais de 0 a 10. As duas equipes também podem gerar apenas partes de proteínas, combinando novas com estruturas já existentes.
A maioria dessas estruturas demonstradas não serviriam para nada na prática. Mas como a função de uma proteína é determinada por sua forma, ser capaz de gerar diferentes estruturas sob demanda é vital.
Criar designs estranhos em um computador é uma coisa. Mas o objetivo é transformá-los em proteínas reais e funcionais. Para testar se o Chroma produziu padrões que poderiam ser feitos realmente, a Generate Biomedicines pegou as sequências de alguns deles, ou seja, as cadeias de aminoácidos que compõem a proteína, e as usou em outro programa de IA. Descobriu-se que 55% das sequências poderiam ser dobradas nos padrões gerados pelo Chroma, o que sugere que estas seriam proteínas viáveis.
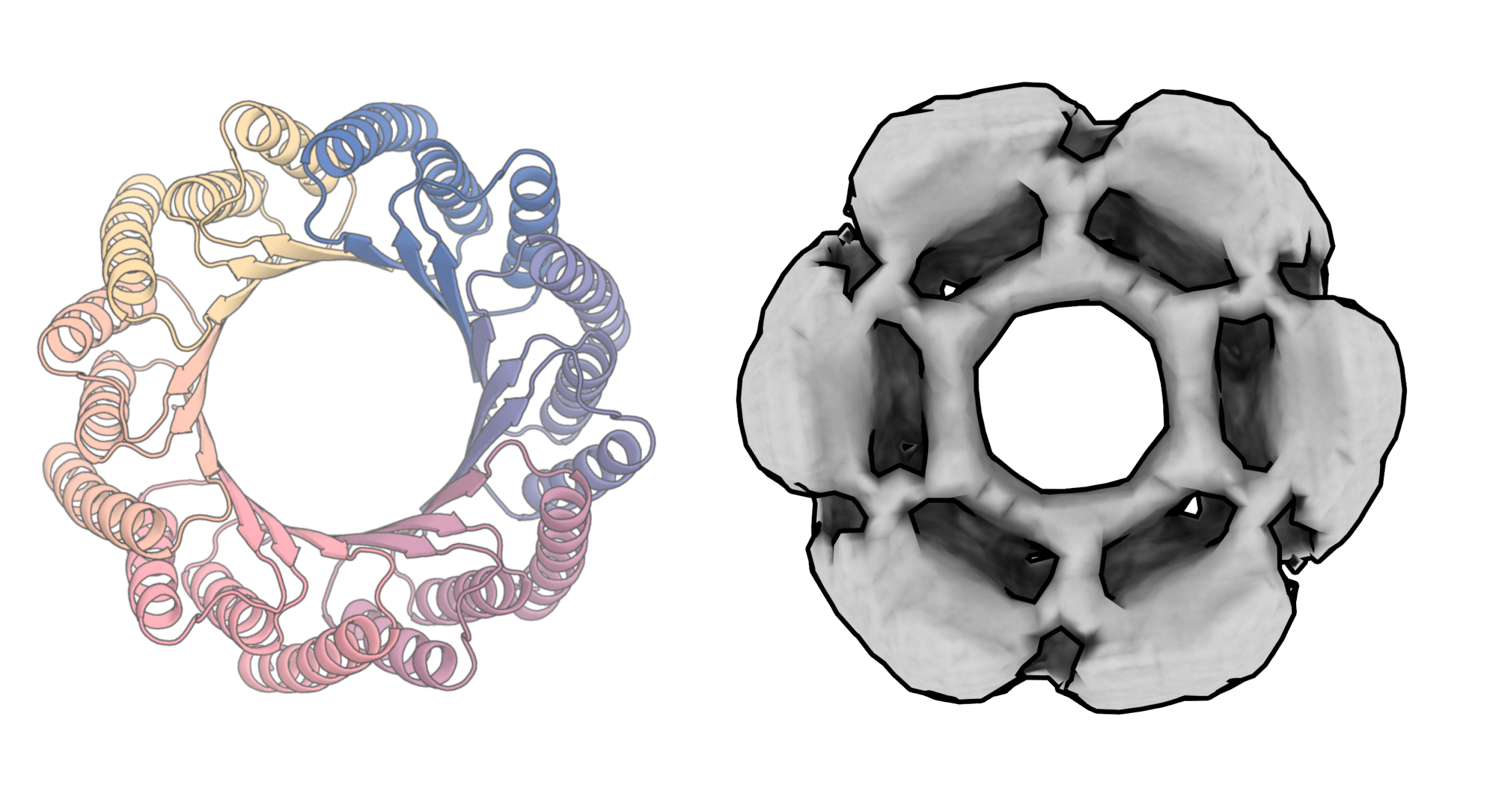
A equipe de Baker fez um teste semelhante. Mas Baker e seus colegas foram muito além da Generate Biomedicines na avaliação de seu modelo. Eles criaram alguns dos padrões da RoseTTAFold Diffusion em seu laboratório. (A Generate Biomedicines diz que também está fazendo testes de laboratório, mas ainda não está pronta para compartilhar os resultados.) “Isso é mais do que apenas uma prova de conceito”, diz Trippe. “Na verdade, estamos usando isso para produzir proteínas realmente funcionais”.
Para Baker, o resultado de destaque é a geração de uma nova proteína que se liga ao paratormônio (PTH), que controla os níveis de cálcio no sangue. “Basicamente, demos ao modelo o hormônio e nada mais e dissemos a ele para produzir uma proteína que se liga-se a ele”, diz ele. Quando testaram a nova proteína no laboratório, descobriram que ela se ligava ao hormônio mais firmemente do que qualquer coisa que pudesse ter sido gerada usando outros métodos computacionais. Até mesmo do que com os medicamentos existentes. “Ele simplesmente criou esse design de proteína do nada”, diz Baker.
Grigoryan reconhece que inventar novas proteínas é apenas o primeiro passo de muitos. “Somos uma empresa farmacêutica”, diz ele. “No final das contas, o que importa é se podemos desenvolver remédios que funcionem ou não”. Medicamentos à base de proteínas precisam ser fabricados em grande número, depois testados em laboratório e, finalmente, em humanos. Isso pode levar anos. Mas ele acha que sua empresa e outras também encontrarão maneiras de acelerar essas etapas.
“A taxa de progresso científico vem crescendo aos trancos e barrancos”, diz Baker. “Mas agora estamos no meio do que só pode ser chamado de revolução tecnológica”.




